Régulation épigénétique via HsMar1
Projet soutenu par le CGO (Cancéropôle du Grand ouest) - axe épigénétique.
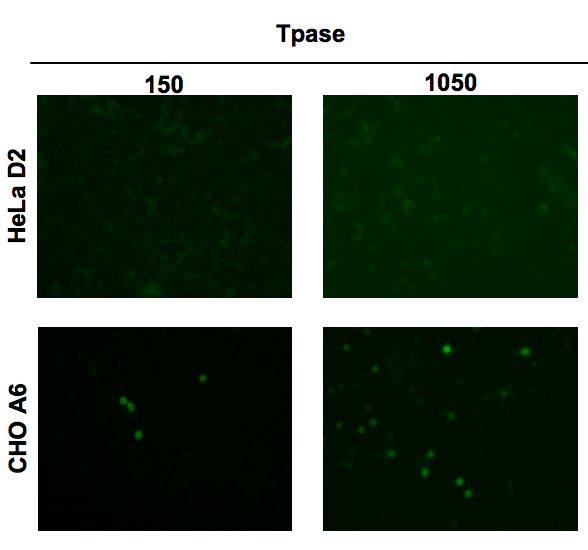
L'équipe de recherche IGC a intégré le transposon HsMar1 dans le génome de cellules HeLa (déjà porteur d’éléments HsMar1, de MITEs d’HsMar1 et d’IR solos) et dans celui de cellules de hamster CHO, où HsMar1 est absent. Les tests d’excision (la première étape de la transposition) ont montré qu’HsMar1 est excisé efficacement du génome des cellules CHO (présence de cellules exprimant la GFP donc vertes) alors qu’il est immobilisé dans le génome des cellules HeLa (absence de cellules vertes), probablement par la régulation exercée par les copies endogènes sur la copie recombinante.
Les marques épigénétiques de cette régulation ont été recherchées notamment en étudiant la méthylation des CpG de la copie HsMar1 recombinante, dans les deux types de cellules (HeLa et CHO). Les séquences situées à côté de l’IR5’ d’Hsmar1 sont methylées dans les cellules HeLa alors qu’elles ne le sont pas dans les cellules CHO, montrant qu’une régulation épigénétique s’installe dans le génome de cellules contenant des copies HsMar1 endogènes.
L'équipe recherche également la présence d’histone H3K9me (marqueur de l’hétérochromatine corrélé à la méthylation de l’ADN) au niveau de la copie recombinante HsMar1 en cellules HeLa, alors que cette modification devrait être absente des copies intégrées dans le génome des cellules CHO. L'étude sera étendue aux copies endogènes complètes d’HsMar1 en cellule HeLa, ainsi qu’aux gènes se situant à proximité de ces copies.
L’objectif est d’identifier des situations pour lesquelles des marques épigénétiques s’étendraient aux gènes adjacents et de valider ainsi l’hypothèse de travail selon laquelle HsMar1 pourrait (par une action en cis) modifier l’expression de ces gènes proches. Parmi ces gènes, l'équipe IGC cherche à trouver ceux impliqués dans la régulation du cycle cellulaire, voire de l’oncogenèse.
